? 2025 最新!Chai AI 多模态基础模型 Chai-1 精准分子结构预测攻略
? 为什么 Chai-1 是结构预测领域的新标杆?
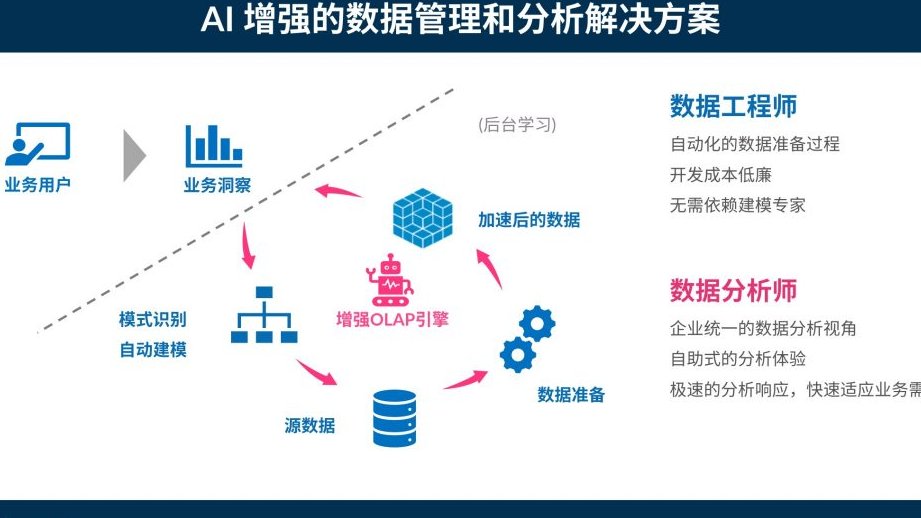
? 多模态融合:让结构预测更「聪明」
?️ 从安装到实战:Chai-1 的保姆级使用指南
Chai-1 提供了 Python 库
chai_lab,安装非常简单。打开终端输入:pip install chai_lab==0.6.1如果需要最新功能,也可以从 GitHub 拉取开发版:
pip install git+https://github.com/chaidiscovery/chai-lab.git注意,模型对计算资源有一定要求。虽然 RTX 4090 也能运行,但推荐使用 NVIDIA A100 80GB 或 H100 80GB GPU,以确保复杂任务的效率。
Chai-1 支持多种输入格式:
- 蛋白质 / 核酸序列:FASTA 文件,每行以
>开头,后跟序列名称和氨基酸 / 核苷酸序列。 - 小分子:SMILES 字符串,例如
CC(=O)O代表乙酸。 - 约束条件:JSON 文件,指定原子间距离、二面角等约束。
最简单的单序列预测命令:
chai-lab fold input.fasta output_folder这条命令会生成 5 个预测模型,默认不使用 MSA 和模板。如果需要提升精度,可以启用服务器端的 MSA 和模板搜索:
chai-lab fold --use-msa-server --use-templates-server input.fasta output_folder对于批量处理或集成到现有工作流,推荐使用 Python API:
from chai_lab.chai1 import run_inference
# 准备输入数据
sequences = ["MKTIIALSYIFCIVLFAYFVDE...", "MAKQLKSLVLLWVLAAQKVG..."]
ligands = ["CC(=O)O", "C1=CC=CC=C1"]
# 执行预测
results = run_inference(sequences, ligands=ligands, constraints="constraints.json")
# 保存结果
for i, pdb in enumerate(results):
with open(f"model_{i}.pdb", "w") as f:
f.write(pdb)
? 深度应用场景:从药物设计到基础研究
在 KRas G12C 抑制剂开发中,Chai-1 准确预测了 AMG 510 与靶点的共价结合模式,RMSD 值仅 0.537 Å,与实验结果高度吻合。用户只需上传蛋白质序列和小分子 SMILES,就能快速获得复合物结构,大大加速了苗头化合物的优化进程。
以 KSI 酶为例,研究人员通过 Chai-1 预测了突变体与非天然底物的结合构象,再结合 Gnina Score 打分,仅通过几个非催化位点的突变就显著提升了底物结合能力。这种「预测 - 突变 - 再设计」的闭环,让酶改造效率提升数倍。
在缺乏完整实验数据时,Chai-1 能仅凭抗原表位的部分接触点信息,生成高可信度的复合物结构。这在新冠中和抗体筛选中已得到验证 —— 模型预测的结合模式与冷冻电镜结果一致,为后续亲和力成熟提供了关键指导。
⚠️ 注意事项与避坑指南
Chai-1 目前仅限非商业用途,学术研究需遵守开源协议。商业用户需联系 Chai Discovery 获取授权。
虽然模型支持单卡运行,但处理长序列或多分子复合物时,内存占用可能急剧增加。建议根据任务规模调整 batch size,或使用分布式训练框架(如 DeepSpeed)优化资源利用率。
尽管 Chai-1 精度很高,但预测结果仍需实验验证。例如,在药物设计中,建议结合分子动力学模拟(如 GROMACS)进一步评估复合物稳定性。
? 与竞品对比:Chai-1 的核心竞争力
| 指标 | Chai-1 | AlphaFold 3 | ESM3-98B |
|---|---|---|---|
| 单序列预测精度 | 0.852 Cα-lddt | 0.843 Cα-lddt | 0.801 Cα-lddt |
| 多聚体预测成功率 | 69.8% (DockQ) | 67.7% (DockQ) | 未公开 |
| 配体预测 RMSD 成功率 | 77% (PoseBusters) | 76% (PoseBusters) | 未公开 |
| 多模态支持 | 序列 + 实验约束 | 序列 + 模板 | 序列 + 结构 |
| 开源程度 | 模型权重 + 代码 | 代码 + 部分权重 | 代码 + 部分权重 |